为了实现细胞类型反卷积和精细空间图谱 (refined spatial map)的构建,2022 年5月2日,美国密歇根大学、生物统计系周翔副教授课题组(https://www.xzlab.org;博士研究生马莹为第一作者)在Nature Biotechnology杂志上发表方法研究型长文(Article):Spatially informed cell type deconvolution for spatial transcriptomics, 开发了CARD,一种对空间转录组数据(spatial transcriptomics)去卷积 (deconvolution)和增强空间分辨率(resolution enhancement)的新方法CARD。

CARD 是一种基于条件自回归(conditional autoregressive;CAR),并结合单细胞测序(single cell RNA-seq;scRNA-seq)数据中细胞类型特异性表达的信息进行反卷积分析的方法。图一显示了CARD的主要步骤。如图所示, CARD 将空间转录组数据和从scRNA-seq构建的细胞类型特异性参考基矩阵(reference basis matrix)通过非负矩阵分解(non-negative matrix factorization)模型结合, 同时依赖于相近位置的细胞类型组成的空间结构相似性的CAR假设。基于细胞类型组成中的邻域相似性,CARD可以进一步用于构建细胞类型组成和基因表达水平的精细空间图。此外,因为相应数据缺失,当reference basis matrix无法从scRNA-seq构建而只有标记基因(marker genes)存在时,CARD 的扩展版本还可以进行无细胞类型特异性参考基矩阵(reference-free)的反卷积分析。这些特性使 CARD 成为一种独特的方法,有助于解开细胞类型的空间定位并促进组织结构的全面映射。CARD方法的更多细节可以在论文中找到(链接)。
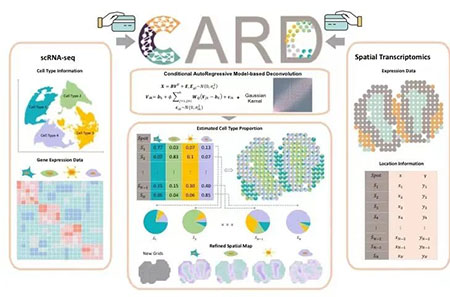
图一. CARD 主要步骤示意图
该课题组通过大量的仿真实验(simulations)和广泛的空间转录组实例数据对CARD进行了评估。其中实例数据包含了生成于不同的物种, 组织结构,空间分辨率和不同的测序技术的空间转录组数据:小鼠嗅球, 人类胰腺癌,小鼠海马和小鼠大脑切片。以人类胰腺癌为例,CARD利用样本匹配的单细胞数据,对人类胰腺癌组织空间转录数据进行反卷积来分解细胞混合物,从而构建细胞类型空间分布图谱, 并确定了两个不同的癌症区域。计算出来每个癌症区域的特征细胞的空间分布也和测出来的标记基因的空间分布相一致。同时,CARD也能精准定位不同细胞类型在不同组织区域的分布, 比如该课题组发现,成纤维细胞(fibroblast cells)和胰腺癌克隆细胞A型(cancer clone A cells)共定位在癌症区域的上部区域, 这可能和成纤维细胞在肿瘤微环境的重要作用有关。而由S100A 基因标记的胰腺癌克隆细胞B型(cancer clone B cells)主要集中分布在癌症区域的底部区域。同时,由CARD构建的细胞类型组成和基因表达的高分辨率图谱使得不同癌症区域之间的区别更加明显。该图谱有助于识别具有不同空间定位的多种细胞类型和分子标记,这些标记定义了进展、异质性和胰腺癌的区室化。此外,CARD基于其他非样本匹配的单细胞数据以及无细胞类型特异性参考基矩阵(reference-free)的反卷积分析得到的细胞类型空间分布图谱也和以上高度一致。
简而言之,该课题组开发了一个快速,高效的反卷积方法CARD。通过数值模拟和实例分析表明该方法可以对细胞类型进行准确的分解, 从而构建高分辨率图谱。CARD的R软件包可以从Zhou Lab 网站https://www.xzlab.org/software.html下载、安装;R软件安装方法及示例数据分析详细说明文档见网站https://github.com/YingMa0107/CARD
原文链接:
https://doi.org/10.1038/s41587-022-01273-7













